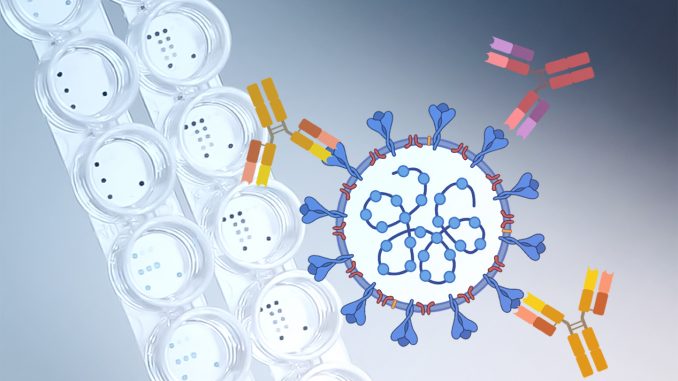
Wissenschaftler des InfectoGnostics Forschungscampus Jena haben einen neuen Microarray entwickelt, mit dem sich Antikörper gegen verschiedene Infektionserreger wie Corona, Masern, Diphterie und Tetanus gleichzeitig nachweisen lassen.
Mit einem einzigen Tropfen Blut des Patienten kann getestet werden, ob das Immunsystem auf eine Impfung angesprochen hat oder eine antikörper-vermittelte Immunität nach Infektion besteht. Für die Entwicklung und Validierung des Tests haben die Campuspartner Leibniz-Institut für Photonische Technologien (Leibniz-IPHT), fzmb GmbH, Virion\Serion GmbH sowie das Universitätsklinikum Jena (UKJ) kooperiert.
Um sich gegen Infektionserkrankungen und Fremdstoffe zur Wehr zu setzen, bildet das Immunsystem des Menschen Antikörper. Doch diese Antikörper richten sich nicht gegen den gesamten Erreger, sondern binden per Schlüssel-Schloss-Prinzip stets an ganz bestimmte molekulare Strukturen des Eindringlings, die man auch „Antigene“ nennt. InfectoGnostics-Wissenschaftler haben nun im RESISTOVAC-Projekt eine Testplattform entwickelt, bei der man Dutzende solcher Antigene auftragen kann. Mit einem einzigen Tropfen Blut des Patienten kann somit parallel auf mehrere Antikörper gegen übliche und neue Infektionskrankheiten getestet werden.
Langzeitgedächtnis gegen Bestandteile eines Erregers
Zentraler Fokus war bei der Entwicklung zunächst ein genaueres Screening der Immunreaktion auf das Coronavirus. Zum Einsatz kam dabei ein sogenannter Protein-Microarray: Das ist ein wenige Millimeter kleiner Chip, auf dem verschiedene Antigene als Fängermoleküle aufgetragen und gebunden sind. Bringt man Antikörper aus dem Patientenblut mit den passenden Antigenen auf dem Chip zusammen, verfärben sich durch eine speziell entwickelte Methode entsprechende Testfelder auf dem Mikroarray – ein gesuchter Antikörper muss also im Blut vorhanden gewesen sein.
Um die Zuverlässigkeit dieses Nachweises zu validieren, haben die InfectoGnostics-Forscher unterschiedliche SARS-CoV-2-Antigene auf den Microarray aufgetragen und gegen reale Proben von Covid-19-Patienten aus Thüringen getestet: „Wir wollten dabei die relativ spät gebildeten IgG-Antikörper nachweisen – eine Art Langzeitgedächtnis des Immunsystems gegen Bestandteile eines Erregers. Für deren Nachweis haben wir 18 Antigen-Kombinationen für das Corona-Virus als Fängermoleküle aufgebracht“, erläutert Sindy Burgold-Voigt, Erstautorin der Studie und Doktorandin am Leibniz-IPHT.
Immunsysteme reagieren äußerst individuell
In ihrer Auswertung stellten die Wissenschaftler fest, dass die Immunantwort individuell sehr verschieden ausfällt: So hatten einige Patienten beispielsweise nur Antikörper gegen ein Protein entwickelt, welches das Erbgut des Virus einhüllt – das Nucleocapsid. Andere Patienten hatten hingegen Antikörper gegen das sogenannte Spike-Protein – die spitzen Strukturen, mit denen sich das Virus an menschliche Zellen anheftet. Wieder andere hatten gegen beide Corona-Antigene die passenden Antikörper.
Antikörper-Abwehr besser verstehen
Die kluge Kombination von verschiedenen Antigenen auf einem einzigen Test kann damit von hoher Bedeutung für das Verständnis der Antikörper-Immunabwehr und die Arbeit an neuen Impfstoffen sein, erläutert Sindy Burgold-Voigt: „Für die Weiterentwicklung und Effizienzanalysen von Impfungen ist es extrem wichtig, dass die Forschung ein diagnostisches Werkzeug hat, mit dem man sich einen schnellen Überblick über die Immunreaktion verschaffen kann.“
Unterstützt wurde die Test-Entwicklung durch das Bundesministerium für Bildung und Forschung im Rahmen des InfectoGnostics-Leitprojekts RESISTOVAC, das die Entwicklung von POC-Tests zur Bestimmung des Immunstatus und bakterieller Resistenzfaktoren zum Ziel hat. Darüber hinaus wurde die Studie durch den Freistaat Thüringen gefördert und durch Mittel der Europäischen Union im Rahmen des Europäischen Sozialfonds (ESF) kofinanziert.
Die Ergebnisse wurden im Fachjournal „Scientific Reports“ publiziert und sind kostenlos zugänglich (DOI: 10.1038/s41598-022-10823-7).
