Wissenschaftler des Heidelberger Instituts für Theoretische Studien (HITS) haben jetzt eine Open-Source Online-Anwendung für die Segmentierung biomedizinischer Aufnahmen entwickelt.
Auf dem Gebiet der bildgebenden Verfahren wurden zuletzt erhebliche Fortschritte erzielt, die zu immer höheren Auflösungen und schnelleren Erfassungszeiten geführt haben. Aufnahmen einzelner Zellen, Gewebeteile und Organe versorgen heutzutage medizinische Fachleute auf der ganzen Welt mit zahlreichen Informationen über den Gesundheitszustand ihrer Patienten. Wie aber lassen sich diese Bilder richtig deuten?
Bisher fehleranfällige Analysen
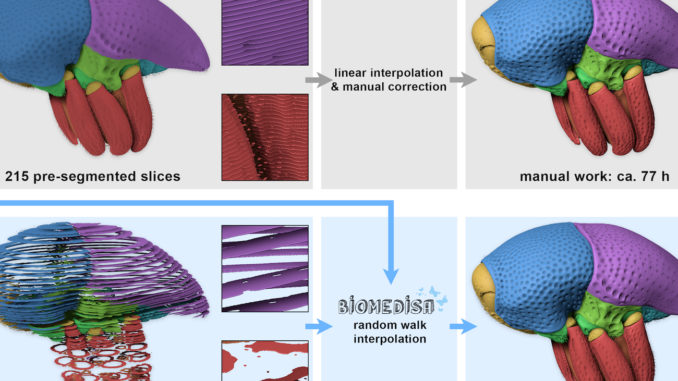
Damit die Aufnahmen ihr Informationspotential entfalten, bedarf es sehr häufig einer manuellen Segmentierung. „Die manuelle Segmentierung unbekannter biomedizinischer Datensätze ist oft sehr zeitaufwändig und fehleranfällig, da sehr viele Schichten segmentiert werden müssen. Zur Analyse dreidimensionaler Bilddaten wird dieses Verfahren aber noch sehr häufig angewendet. In vielen Instituten ist es tatsächlich gang und gäbe, Heerscharen von speziell geschulten Studierenden extra für diese Aufgabe einzusetzen,” so Philipp Lösel aus der Forschungsgruppe „Data Mining and Uncertainty Quantification” (DMQ) am HITS, der Biomedisa entwickelt hat.
Schnellere und genaue Segmentierung
Die Biomedical Image Segmentation App „Biomedisa“ (https://biomedisa.org) ist eine frei verfügbare und leicht zu bedienende Open-Source Online-Anwendung, die speziell für die halbautomatische Segmentierung entwickelt wurde. Die Segmentierung basiert dabei auf einer intelligenten Interpolation einiger weniger vorsegmentierter Schichten, wobei die zugrundeliegenden Bilddaten vollständig mit einbezogen werden. Dadurch können wesentlich größere Abstände zwischen den Schichten gelassen werden. „Biomedisa kann den Segmentierungsprozess enorm beschleunigen und liefert gleichzeitig genauere Ergebnisse als dies bei einer rein manuellen Segmentierung der Fall ist,” so Thomas van de Kamp, Biologe am KIT, der aus eigener Erfahrung weiß, wie mühevoll eine manuelle Bildsegmentierung sein kann.
Die Plattform ist über einen Webbrowser zugänglich (https://biomedisa.org), eine komplexe und langwierige Konfiguration der Software oder der Modellparameter ist nicht erforderlich. Die One-Button-Lösung kann bei verschiedenen 3D-Bildgebungsverfahren und zahlreichen biomedizinischen Anwendungen eingesetzt werden.
Die Arbeit fand im Rahmen eines interdisziplinären Forschungsprojektes mit Wissenschaftler/-innen der Universität Heidelberg, dem Karlsruher Institut für Technologie (KIT) und der Technischen Universität Darmstadt statt. Die Ergebnisse , die sich vor allem an Forschende ohne fundierte Computerkenntnisse richten, wurden jetzt in der Fachzeitschrift Nature Communications veröffentlicht.
Das Heidelberger Institut für Theoretische Studien (HITS) wurde 2010 von SAP-Mitbegründer Klaus Tschira (1940-2015) und der Klaus Tschira Stiftung als private, gemeinnützige Forschungseinrichtung ins Leben gerufen. Das HITS betreibt Grundlagenforschung in den Naturwissenschaften, der Mathematik und der Informatik. Die Forschungsfelder reichen von der Molekularbiologie bis zur Astrophysik.
